Análisis Geoestadístico de la Vigilancia Entomológica con Ovitrampas en INLA
eggs_prediction_hotspots.RmdVigilancia Entomológica con Ovitrampas en México
Ovitrampa
Una ovitrampa consiste en un vaso de plástico negro de una capacidad de un litro cubierto en su superficie interior con papel pellón (F-1600) y rellenado con agua potable aproximadamente 3/4 ó hasta 2-cm antes su borde superior.
Sistema de Vigilancia Entomológica con Ovitrampas.
La vigilancia entomológica con ovitrampas consiste en instalar 4 ovitrampas por manzana (una ovitrampa por cada lado de la manzana) con un intervalo de separación de cada 4 a 6 manzanas en zonas planas y con un intervalo de separación de 2 a 3 manzanas en zonas con relieve.
deneggs::blocks_surveillance(locality = "Ticul",
cve_edo = "31")Semanalmente a las ovitrampas instaladas en lugares sombreados a una altura de 1.5, se les retira el sustrato de oviposición y se les cambia por un sustrato nuevo y agua. El número de huevos en la papeleta es contado y registrado. La información asociada a la papeleta (localidad, municipio, manzana, sector, fecha, colector y número de huevos) es capturado en el sistema de Monitoreo Integral de Vectores de la Secretaría de Salud Federal (http://geosis.mx/Aplicaciones/SisMV/Home/Dengue) (Secretaría de Salud [SSA], 2019; Mejía-Guevara et al, 2020).
Análisis Geoestadístico
Los datos del registro del número de huevos generados de la vigilancia entomológica con ovitrampas (es decir el número de huevos con las coordenadas geográficas finitas en la localidad de estudio), es conceptualizado para fines del análisis espacial como una realización parcial de un proceso espacial continuo de todo el dominio de estudio (localidad). Las características y propiededades de esta realización parcial permiten conocer todo el proceso espacial y en consecuencia realizar predicciones del número de huevos en las áreas (manzanas sin muestrear) donde no se instalaron ovitrampas.
Para predecir el número de huevos en toda la localidad, se asume que la variable de respuesta $\color{#2EB67D}{y_i}$ tiene una distribución discreta para los datos de conteo (binomial negativa o cero inflado binomial negativa) y que existe un efecto espacial $\color{#ECB22E}{u{_s{_i}}}$ definido por cada suscripto como la longitud y la latitud de la ovitrampa 1 a la n en cada localidad de estudio. Las $\color{#ECB22E}{u{_s{_i}}}$ son espacialmente correlacionadas (existe dependencia espacial) con media y una matriz de covarianzas Σ. La solución del efecto espacial es resuelta a través de Stochastic Partial differential Equation (SPDE) e Integrated Nested Laplace Approximations (INLA). El modelo es parametrizado con la ecuación $\color{#2EB67D}{y_i} = \color{#36C5F0}{\beta_0} + \color{#ECB22E}{u{_s{_i}}} +\color{#6200B4}{e_i}$ y las predicciones del número de huevos en la localidad fueron utilizados para calcular los hotspots de las abundancias de huevos de Ae. aegypti en la localidad.
Análisis Geoestadístico de la Vigilancia Entomológica
En analisis geoestadístico de la vigilancia entomológica con ovitrampas se realiza con el paquete deneggs. El paquete es parte del dengueverse & tiene tres funciones para realizar la predicción del número de huevos en áreas donde no se colectó de una localidad.
La función spde_pred_map tiene la característica de realizar el analisis geoestadístico por semana con siete diferentes distribuciones (poisson, zeroinflatedpoisson0, zeroinflatedpoisson1, nbinomial, nbinomial2, zeroinflatednbinomial0, & zeroinflatednbinomial1). La función eggs_hotspots realiza el análisis por semana con solo una distribución y la función eggs_hotspots_week realiza el análisis para todas las semanas del año. Los argumentos estas tres funciones son definidos en la siguiente tabla.
Predicción de huevos por semana
función spde_pred_map
Código
# Step 1. define the path ####
path_ovitraps <- "/Users/fdzul/Dropbox/CENAPRECE/2024/12_guerrero"
path_coordinates <- paste(path_ovitraps,
"DescargaOvitrampasMesFco.txt",
sep = "/")
# Step 2. run the function ####
library(magrittr)
library(sf)
x <- deneggs::spde_pred_map(path_lect = path_ovitraps,
path_coord = path_coordinates,
cve_ent = "12",
locality = "Acapulco de Juarez",
longitude = "Pocision_X",
latitude = "Pocision_Y",
aproximation = "gaussian",
integration = "eb",
k = 30, plot = TRUE, week = 30,
var = "Huevecillos",
cell_size = 2000, alpha = 0.99,
palette_vir = "magma",
leg_title = "Huevos")
Elementos
names(x)
#> [1] "data" "mesh" "map" "mod" "loc" "dics" "hotspots"Distribuciones
# x$dics
x$dics |>
dplyr::arrange(dic)
#> dic fam
#> 1 43202.56 zeroinflatednbinomial1
#> 2 43257.58 zeroinflatednbinomial0
#> 3 43281.95 nbinomial
#> 4 43363.10 zeroinflatedpoisson0
#> 5 43429.88 zeroinflatedpoisson1
#> 6 44009.80 nbinomial2
#> 7 54869.26 poissonMapa

función eggs_hotspots
Código
path_ovitraps <- "/Users/fdzul/Dropbox/CENAPRECE/2025/27_tabasco"
path_coord <- paste(path_ovitraps,"DescargaOvitrampasMesFco.txt",
sep = "/" )
x <- deneggs::eggs_hotspots(path_lect = path_ovitraps,
cve_ent = "27",
locality = "Villahermosa",
path_coord = path_coord,
longitude = "Pocision_X",
latitude = "Pocision_Y",
aproximation = "gaussian",
integration = "eb",
fam = "zeroinflatednbinomial1",
k = 80, palette_vir = "magma",
leg_title = "Huevos", plot = FALSE,
hist_dataset = FALSE, sem = 29,
var = "eggs", cell_size = 3000,
alpha = .99)Elementos
names(x)
#> [1] "data" "map" "loc" "rast_loc" "mod" "mesh" "dics"
#> [8] "hotspots"Mapa
x$map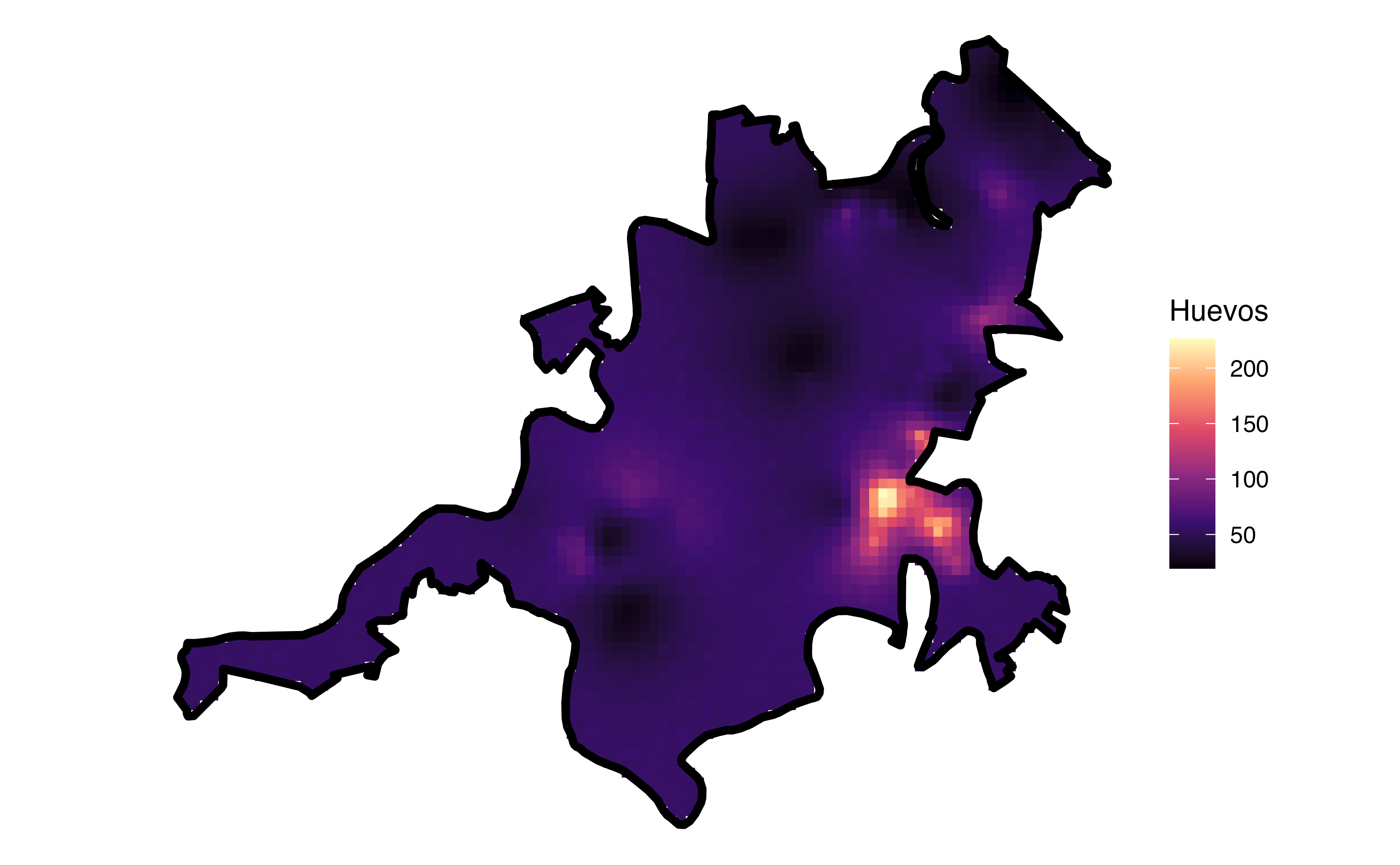
Predicción de huevos por año
función eggs_hotspots_week
Código
# Step 1. define the path ####
path_ovitraps <- "/Users/fdzul/Dropbox/CENAPRECE/2025/11_guanajuato"
path_coordinates <- paste(path_ovitraps, "DescargaOvitrampasMesFco.txt", sep = "/")
library(sf)
library(INLA)
# # Step 2. run the spde model of Leon ####
leon <- deneggs::eggs_hotspots_week(cve_mpo = "020",
cve_edo = "11",
year = "2024",
hist_dataset = FALSE,
locality = "Leon de Aldama",
path_vect = path_ovitraps,
path_coord = path_coordinates,
integration_strategy = "eb",
aproximation_method = "gaussian",
fam_distribution = "zeroinflatednbinomial1",
kvalue = 40,
plot = FALSE,
palette.viridis = "viridis",
cell.size = 2000,
alpha.value = .99)Mapa de Intensidad
deneggs::map_eggs_hotspots(betas = leon$betas,
locality = "Leon de Aldama",
cve_edo = "11",
palette = rcartocolor::carto_pal,
name = "SunsetDark",
static_map = FALSE)